
Os resíduos plásticos tornaram-se recentemente uma grande preocupação ambiental global e um dos maiores desafios tem sido a busca por opções alternativas de gerenciamento. Vários estudos revelaram o potencial de várias espécies de coleópteros para degradar plásticos, e este é o primeiro trabalho de pesquisa sobre o potencial de degradação do plástico por larvas de farinha menores da África. Este estudo avaliou todo o perfil mitogenômico da larva da farinha menor para identificar melhor o inseto. A capacidade da larva da farinha de consumir poliestireno (PS) também foi avaliada juntamente com sua diversidade de microbiota intestinal associada. Nossos resultados mostraram um genoma mitocondrial circular completo que se agrupou de perto no gênero Alphitobius, mas também sugeriu que nosso inseto pode ser uma nova subespécie que requer identificação adicional. Durante os testes de alimentação PS, as taxas de sobrevivência global das larvas diminuíram quando alimentadas com uma dieta PS única, enquanto a ingestão de PS aumentou em um período de 30 dias. As bactérias predominantes observadas nas larvas alimentadas com dietas PS foram Kluyvera, Lactococcus, Klebsiella, Enterobacter e Enterococcus, enquanto Stenotrophomonas dominou a dieta controle. Esses achados demonstraram que a recém-identificada larva da farinha pode sobreviver com uma dieta PS e tem um consórcio de bactérias importantes fortemente associadas à degradação PS. Este trabalho fornece uma melhor compreensão das aplicações de biorremediação, abrindo caminho para mais pesquisas sobre as vias metabólicas de micróbios que degradam o plástico e trazendo esperança para resolver a poluição por resíduos plásticos, ao mesmo tempo em que fornece proteína de inseto de alto valor para uma economia circular.
Introdução
O acúmulo de resíduos plásticos tornou-se uma grande preocupação ambiental global que está aumentando rapidamente e a África não é exceção. A produção global de plásticos está projetada para atingir 460 milhões de toneladas, com volumes de produção que devem continuar aumentando nas próximas décadas1. Até 2050, estima-se que a produção atinja 590 milhões de toneladas, indicando um crescimento de mais de 30%2. A África se tornou o segundo continente mais poluído do mundo, com mais de 500 contêineres de resíduos sendo importados a cada mês, e estima-se que esse valor dobre nos próximos 5 anos3. Além disso, a taxa atual de reciclagem de resíduos plásticos é de apenas 14 a 18% e essa baixa taxa de reciclagem, juntamente com a natureza duradoura dos plásticos e microplásticos, levou a efeitos prejudiciais ao meio ambiente, contribuindo para as mudanças climáticas, além de representar riscos para a saúde humana4.
O poliestireno (PS) é um polímero barato e durável cuja produção anual é estimada em 20 milhões de toneladas métricas5. O poliestireno é um dos principais microplásticos que se acumulam tanto nos ecossistemas terrestres quanto aquáticos6. Os resíduos de poliestireno resultam do uso comercial de sua forma comum, o isopor, que é usado para várias aplicações, incluindo recipientes de armazenamento de alimentos, embalagens de equipamentos, pratos e copos descartáveis e isolamento na construção7. Para combater essa crise crescente, é necessário dar mais atenção a vários métodos de degradação do poliestireno sem poluição secundária. Diferentes métodos têm sido usados para reciclar PS, incluindo métodos químicos, térmicos e mecânicos8. No entanto, esses métodos são caros e produzem compostos aromáticos que podem ampliar os impactos ambientais negativos9. Estudos recentes reorientaram os interesses de pesquisa sobre a capacidade de insetos como T. molitor, G. mellonella e Z. atratus e seus micróbios de biodegradar o PS como uma estratégia sustentável promissora10,11,12,13. Recentemente, uma nova espécie de larva de farinha denominada larva de farinha escura foi criada no Centro Internacional de Fisiologia e Ecologia de Insetos (icipe), no Quênia. As características desta nova espécie são semelhantes às de outras larvas de farinha da família Tenebrionidae e especificamente relacionadas com a subfamília Alphitobiini. As larvas de farinha menores são consideradas uma praga importante na produção de aves e são facilmente observadas em cama de frango, galinheiros e esterco14. Esta espécie causa danos aos aviários e pode ser um reservatório para patógenos avícolas. No entanto, um estudo recente sobre a larva da farinha menor, Alphitobius diaperinus, concluiu que esta espécie é capaz de ingerir e degradar PS e identificou bactérias Cronobacter, Pseudomonas e Kocuria que foram propostas como tendo capacidade de degradação de plástico15.
Várias espécies de lepidópteros, como a mariposa indiana (Corcyra cephalonica) e a mariposa da cera menor (Achroia grisella), demonstraram a capacidade de degradar plásticos de baixa densidade16,17,18. No entanto, os tenebroinídeos são uma das espécies de insetos degradadores de plástico mais caracterizadas, e suas bactérias intestinais forneceram informações valiosas sobre os papéis que os micróbios intestinais desempenham na degradação do plástico11. Conforme descrito recentemente por19, as larvas de farinha amarelas foram capazes de sobreviver com dietas alimentadas com PS. Os autores documentaram a capacidade das larvas de farinha de degradar o PS com aproximadamente 47,7% do isopor ingerido e convertido em dióxido de carbono, enquanto o resíduo foi excretado como excremento. Nos experimentos subsequentes20, demonstraram os papéis distintos que a microbiota intestinal desempenha no processo de biodegradação, onde estabeleceram que Exiguobacterium sp. a cepa YT2 isolada do intestino da larva da farinha foi capaz de formar um biofilme ao redor do PS e degradar o PS. Outras espécies de insetos, como a mariposa-da-cera (Galleria mellonella) e os super vermes (Zophobas morio), também foram identificadas como tendo as mesmas habilidades que as larvas de farinha para degradar o plástico e convertê-lo em compostos não perigosos11. Em um estudo feito por12, as capacidades de degradação de PS de T. molitor, G. mellonella e Z. atratus foram comparadas e relataram que todas as três espécies foram capazes de degradar PS em diferentes graus, com os super vermes tendo as maiores taxas de consumo de PS, enquanto a larva de farinha amarela despolimerizou o PS fortemente. Além disso, eles identificaram bactérias Enterococcus e Enterobacteriaceae em todas as três espécies que podem desempenhar um papel na degradação do plástico.
No entanto, faltam informações sobre a caracterização e identificação de potenciais bactérias e fungos degradadores de PS no intestino de larvas de farinha menores, particularmente na África. Portanto, o escopo da pesquisa atual foi investigar a modulação da microbiota intestinal da recém-identificada larva da farinha escura em resposta a dietas alimentadas com PS, bem como a identificação por meio do sequenciamento do genoma mitocondrial completo. Isso lançará luz sobre a possibilidade de a larva da farinha endêmica degradar o PS e os micróbios associados que contribuem para sua eficácia. Esta pesquisa estabelece as bases para estudos futuros sobre a reciclagem microbiana de resíduos plásticos em biomassa rica em proteína de inseto de alto valor para ração animal.
Metodologia
Criação de larvas de farinha
A colônia-mãe das larvas de farinha menores foi criada na unidade de criação e contenção de animais (ARCU) do Centro Internacional de Fisiologia e Ecologia de Insetos (icipe). As larvas foram criadas em bandejas (46W × 60L × 10H cm) providas de cama de farelo de trigo como fonte nutricional. O farelo de trigo usado em testes de criação e alimentação foi adquirido da PEMBE Feeds LTD, Nairóbi. As larvas também receberam resíduos de hortaliças e frutas como fonte de água durante todo o período de criação. A sala de criação foi mantida a uma temperatura de 26 ± 2 °C e 50–70 ± 2% de umidade com iluminação normal e aeração adequada. Da colônia-mãe, larvas de 6º ínstar foram coletadas para testes de alimentação e posterior trabalho molecular, conforme descrito abaixo.
Sequenciamento, montagem e anotação do mitogenoma
Amostras de duas larvas de farinha menores adultas foram usadas para recuperar o perfil mitogenômico completo e o DNA genômico completo isolado de cada amostra usando o kit de extração genômica Isolate II (Bioline, Londres, Reino Unido) seguindo as instruções do fabricante. O DNA extraído foi então enviado para a BGI Genomics (BGI, Tai Po, NT, Hong Kong) para sequenciamento do genoma completo usando a plataforma de sequenciamento DNBseq. O mapeamento e montagem dos mitogenomas a partir dos contigs sequenciados foi feito usando o pipeline MitoZ v2.421. Em resumo, o MitoZ filtra as leituras brutas e, em seguida, monta o mitogenoma com base em uma versão modificada do SOAPdenovo-Trans. Usamos o assembly de modo rápido padrão, onde o MitoZ filtrou as sequências do mitogenoma candidatas usando uma pontuação de confiança e anotou os PCGs, tRNAs e rRNAs. O servidor web MITOS foi usado para comparar as anotações para garantir a anotação correta de todos os genes e também usado para prever as estruturas do tRNA. A composição de nucleotídeos, AT e GC foram calculadas usando primo geneoso. A relação filogenética foi inferida para 21 mitogenomas da família Tenebrionidae, incluindo nossa própria larva de farinha escura, e o alinhamento de múltiplas sequências foi usado para construir uma árvore filogenética de junção de vizinhos usando o parâmetro Kimura 2 no MEGA22.
Consumo de PS e taxas de sobrevivência
Os materiais de poliestireno (PS) utilizados para os ensaios de alimentação foram coletados do isopor utilizado nas embalagens dos equipamentos armazenados no icipe. Para avaliar o consumo de PS, as larvas de 6º ínstar foram colocadas em recipientes de armazenamento de alimentos (H110 × W208 × L208 mm) em sala escura mantida a temperatura constante de 26 ± 2 °C e 70 ± 2% de umidade. Três dietas experimentais foram comparadas por 30 dias: farelo isolado (3,6 g), PS isolado (3,6 g) e farelo + PS (3,6 g:3,6 g). A relação acumulada de farelo para PS (B: PS) foi de 1:1 g/g. Os experimentos foram replicados 3 vezes com 100 larvas em cada dieta de tratamento. O experimento foi conduzido até o início da pupação. A cada 5 dias, as larvas foram contadas e as larvas mortas removidas e o PS residual das dietas PS foi pesado. As taxas de sobrevivência foram calculadas como a porcentagem de larvas vivas com base no número inicial de cada dieta de tratamento (n = 100). As taxas de consumo de PS foram estimadas como o peso do PS consumido dividido pelo peso inicial do PS (3,6 g). Esta metodologia foi adaptada de um estudo anterior realizado por20.
Análise da comunidade microbiana
Após 30 dias, 5 larvas de cada recipiente foram coletadas aleatoriamente e todo o conteúdo intestinal foi colhido de cada larva. As entranhas foram separadas em 3 partes (intestino anterior, intestino médio e intestino posterior). Seguindo as instruções do fabricante, o DNA genômico foi extraído de cada amostra intestinal usando o kit de extração genômica Isolate II (Bioline, Londres, Reino Unido). Um espectrofotômetro nanodrop 2000/2000c (Thermo Fischer Scientific, Wilmington, EUA) foi usado para determinar a concentração e a qualidade do DNA, e aqueles com boa qualidade com uma faixa de A260nm/A280nm de 1,8–2,0 foram escolhidos para análises de metabarcode. O gene 16S rRNA bacteriano de comprimento total de ~ 1500 pb foi sequenciado usando um dispositivo MinION da Oxford Nanopore Technologies (ONT) com células de fluxo R9.4.1. As bibliotecas foram preparadas com o kit de código de barras 16S SQK-16S024 de acordo com as instruções do fabricante. Os seguintes componentes foram utilizados na preparação da biblioteca com etapa de PCR: 10 pmol L−1 de cada código de barras 16S, 10 ng L−1 do molde do ADN, 0.625 U L−1 MyTaq DNA polimerase (Bioline) e tampão de reação 5X MyTaq (5 mM dNTPs, 15 mM MgCl2, estabilizador e intensificador) (Biolinha). As reações foram executadas em um termociclador Master cycler Nexus gradient (Eppendorf, Alemanha) sob as seguintes condições: desnaturação inicial por 2 min a 95 °C, seguida por 40 ciclos de desnaturação por 30 s a 95 °C, recozimento por 45 s a 55 °C, extensão por 1 min a 72 °C e uma etapa final de extensão de 10 min a 72 °C. As bibliotecas com código de barras foram purificadas usando contas AMPure XP (Agencourt Bioscience) e agrupadas antes de serem carregadas nas células de fluxo para sequenciamento.
Sequenciamento e análise de dados
O sequenciamento foi feito por 4 h com chamada de base ao vivo no software MinKNOW (v 20.10.3) na nuvem ONT23. As leituras que passaram no sequenciamento geraram arquivos fastq que foram analisados usando o pipeline q2ONT (https://github.com/DeniRibicic/q2ONT) que usa recursos incorporados no QIIME224. Neste pipeline, todos os arquivos fastq foram concatenados e leituras demultiplexadas em seus respectivos códigos de barras usando a ferramenta Porechop (v 0.2.4) (https://github.com/rrwick/Porechop). As leituras demultiplexadas foram então cortadas e os adaptadores removidos para um comprimento mínimo de 1000 bp usando a ferramenta trimmomatic v 0,3925. As sequências quiméricas foram verificadas usando VSEARCH e filtradas usando a ferramenta UCHIIME. As leituras foram então alinhadas ao MAFFT para gerar uma árvore enraizada e a taxonomia foi atribuída usando o banco de dados SILVA132. O agrupamento da Unidade Taxonômica Operacional (OTU) foi realizado usando a abordagem de referência aberta com 85% de similaridade. O software R foi então usado para a análise a jusante e, para isso, todas as saídas QIIME2 foram importadas para R. OTUs não atribuídas a nenhuma taxonomia foram filtradas e aquelas que foram atribuídas foram usadas para gerar gráficos de barras cumulativas dos gêneros mais abundantes presentes. Os parâmetros de diversidade alfa e beta foram calculados usando o pacote Phyloseq em R26. As estimativas de diversidade alfa foram calculadas usando o índice de Shannon, enquanto a diversidade beta foi calculada usando o método de distância de dissimilaridade de Bray-Curtis.
Análise estatística
R (versão 4.0.2) para executar todas as análises estatísticas27. A ANOVA foi utilizada para analisar as diferenças no consumo de dieta e nas taxas de sobrevida, seguida de comparação pareada usando o teste t de Student com correção de Tukey para analisar as diferenças entre as dietas28. PERMANOVA foi usado para comparar as diferenças estatisticamente significativas entre as comunidades microbianas nos 3 grupos de dieta usando a função Adonis em R usando 999 permutações.
Resultados
Sequenciamento, mapeamento e montagem do mitogenoma completo da larva da farinha africana
O sequenciamento do mitogenoma DNBseq resultou em cerca de 14 milhões de leituras que foram usadas para montar e anotar o genoma mitocondrial completo da larva da farinha escura (Tenebrionidae: Alphitobiini). O mitogenoma circular tinha 15.509 pb e a orientação e ordem dos genes eram idênticas a outros mitogenomas disponíveis publicamente para espécies da família Tenebrionidae. Os mitogenomas de tenebrionídeos revelaram estruturas genômicas altamente conservadas, como ordem gênica, conteúdo de nucleotídeos e composição de aminoácidos, quando comparados aos estudos publicados sobre larvas de farinha na literatura. Consistia em 22 tRNAs, 13 genes codificadores de proteínas (PCGs) e 2 rRNAs (Tabela S1). A fita principal (fita J) carregava 23 genes (9 PCGs e 14 tRNAs), enquanto a fita N menor carregava 14 genes (4 PCGs, 8 tRNAs e 2 rRNAs) (Fig. 1). O comprimento médio combinado de todos os 13 PCGs foi de 10.964, que variou entre 1798 (ND5) e 156 (ATP8) (Tabela S2).
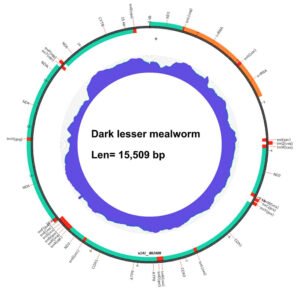
Figura 1: Um mapa circular ilustrando a ordem dos genes do mitogenoma da larva da farinha menor.
O rRNA 16S (1307 pb) estava localizado entre o tRNAL e tRNAV e o rRNA 12S (767 pb) estava localizado entre o tRNAV e a região rica em AT. A maior região não codificante (886 pb) foi anotada como a região rica em AT, localizada entre o rRNA 12s e o tRNAIle (Fig. 1).
Os 22 genes de tRNA foram interespaçados em toda a região codificante e os tamanhos variaram entre 60 e 70 pb (Tabela S1). Todos os tRNAs foram dobrados em uma estrutura típica de folha de trevo, exceto o tRNASor que não tinha um braço de di-hidroudina (DHU) (Fig. S1). O mitogenoma apresentou alto teor de A + T típico de insetos.
A árvore filogenética mostrou que o mitogenoma se agrupou com Alphitobius sp. da tribo Alphitobiini e mostrou que esta tribo se agrupou intimamente com as tribos Helopini e Blaptini (Fig. 2).
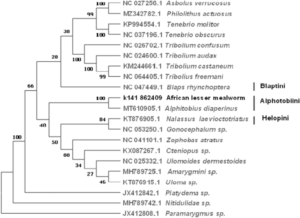 Figura 2: Árvore filogenética de máxima verossimilhança de 21 genomas mitocondriais de Tenebrionidae. A árvore filogenética foi baseada no parâmetro Kimura 2 com 1000 repetições de bootstrap.
Figura 2: Árvore filogenética de máxima verossimilhança de 21 genomas mitocondriais de Tenebrionidae. A árvore filogenética foi baseada no parâmetro Kimura 2 com 1000 repetições de bootstrap.
Consumo de poliestireno e taxas de sobrevivência
A larva da farinha menor foi capaz de mastigar, cavar e se alimentar com sucesso dos blocos de isopor (Fig. 3). Houve aumento progressivo do consumo de SP ao longo do período de 30 dias. Dos 3,6 g iniciais de isopor, o consumo total resultante ao final do experimento foi de 11,7% por todas as larvas de farinha alimentadas com as dietas PS. A co-alimentação das larvas de farinha com farelo e PS aumentou as taxas de consumo de PS em comparação com aquelas fornecidas com dietas de PS solitárias (Fig. 4). As taxas de sobrevivência de larvas de farinha em todos os três grupos de tratamento foram superiores a 80% durante todo o período de alimentação. No entanto, a taxa de sobrevivência das larvas de farinha diminuiu significativamente quando as larvas de farinha foram alimentadas apenas com a dieta PS (Fig. 5A). O consumo de PS aumentou, à medida que as taxas de sobrevivência diminuíram (Fig. 5B).
 Figura 3: Bloco de isopor antes da alimentação (A), Bloco de isopor após 30 dias de alimentação e consumo do poliestireno (PS) evidente pelos orifícios e túneis formados (B), larvas de farinha alimentando-se de poliestireno e farelo (C), larvas de farinha alimentando-se de dieta de poliestireno (D).
Figura 3: Bloco de isopor antes da alimentação (A), Bloco de isopor após 30 dias de alimentação e consumo do poliestireno (PS) evidente pelos orifícios e túneis formados (B), larvas de farinha alimentando-se de poliestireno e farelo (C), larvas de farinha alimentando-se de dieta de poliestireno (D).
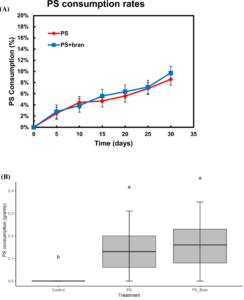
Figura 4: As taxas de consumo de PS durante o período de alimentação de 30 dias (A), Box plot mostrando que não houve diferenças significativas nas taxas de consumo de PS entre as dietas de PS (B).
 Figura 5: A probabilidade de sobrevivência (A) e (B) da eficiência de consumo de PS em comparação com as taxas de sobrevivência no período de 30 dias.
Figura 5: A probabilidade de sobrevivência (A) e (B) da eficiência de consumo de PS em comparação com as taxas de sobrevivência no período de 30 dias.
Comunidades microbianas intestinais
Após o sequenciamento, um total de 300.062 leituras e 428 OTUs foram identificadas. Os filos predominantes identificados no intestino de larvas de farinha submetidas a diferentes tratamentos dietéticos incluíram Proteobacteria e Firmicutes, mas em menor grau Cyanobacteria, Actinobacteria e Bacteroidetes. No nível de classe, γ-proteobactérias e bacilos foram os mais abundantes. O agrupamento de OTUs no nível de família mostrou 4 famílias predominantes: Enterobacteriaceae, Streptococcaceae, Xanthomonadaceae e Enterococcaceae (Fig. S2). Uma análise comparativa mais aprofundada mostrou que as bactérias do gênero Kluyvera, Lactococcus, Stenotrophomonas, Klebsiella, Enterococcus e Citrobacter foram dominantes em todos os 3 grupos de dieta (Fig. 6A). Kluyvera, Lactococcus e Klebsiella foram mais abundantes nas amostras intestinais de larvas de farinha alimentadas exclusivamente com PS. As bactérias predominantes de larvas de farinha fornecidas com uma dieta de farelo de trigo único foram Staphylococcus e Stenotrophomonas (Fig. 6B). As seções superior e média das larvas de farinha alimentadas com farelo de trigo foram dominadas por Stenotrophomonas, enquanto o intestino posterior foi dominado por Staphylococcus. As larvas alimentadas com dietas combinadas de PS e farelo de trigo foram dominadas por Lactococcus nas seções do intestino superior e Kluyvera no intestino grosso. Para as larvas alimentadas exclusivamente com a dieta PS, a seção do intestino grosso foi dominada por Enterococcus e Citrobacter, enquanto Stenotrophomonas foi comum no intestino superior (Fig. 6C).
 Figura 6: Abundância relativa bacteriana nos 3 tratamentos dietéticos. (A) O gráfico de barras empilhadas mostrou abundância bacteriana no nível do gênero, (B) gráfico circos mostrando os 14 principais gêneros de bactérias abundantes e (C) abundância bacteriana cumulativa no nível do gênero das seções anterior, média e posterior das larvas de farinha alimentadas com as 3 dietas.
Figura 6: Abundância relativa bacteriana nos 3 tratamentos dietéticos. (A) O gráfico de barras empilhadas mostrou abundância bacteriana no nível do gênero, (B) gráfico circos mostrando os 14 principais gêneros de bactérias abundantes e (C) abundância bacteriana cumulativa no nível do gênero das seções anterior, média e posterior das larvas de farinha alimentadas com as 3 dietas.
O diagrama de Venn revelou OTUs únicas da larva da farinha quando alimentada com os três tratamentos dietéticos (Fig. 7). A exposição de larvas de farinha a dietas à base de PS aumentou a riqueza da diversidade bacteriana do intestino em comparação com aquelas em dietas de controle. Das 428 OTUs identificadas, apenas 49 táxons foram compartilhados entre os 3 grupos de dieta. As dietas PS tiveram 58 OTUs compartilhadas, que foram altas em comparação com aquelas compartilhadas com a dieta controle. A dieta PS e farelo de trigo apresentou o maior número de táxons únicos, seguida pela dieta somente PS. O controle que possuía farelo teve apenas o menor número de OTUs identificadas (Fig. 7).
 Figura 7: Diagrama de Venn representando os táxons de bactérias compartilhadas de larvas de farinha alimentadas nas 3 dietas com os táxons únicos mostrados em cada grupo.
Figura 7: Diagrama de Venn representando os táxons de bactérias compartilhadas de larvas de farinha alimentadas nas 3 dietas com os táxons únicos mostrados em cada grupo.
A diversidade alfa medida usando o índice de Shannon não variou significativamente entre os vários tratamentos dietéticos. Aqueles alimentados com rações à base de PS mostraram uma comunidade mais diversificada do que aqueles submetidos a dietas de farelo de trigo (Fig. 8). O gráfico PCoA baseado na matriz de dissimilaridade de Bray Curtis e a análise PERMANOVA confirmaram essa semelhança. Como mostrado na Fig. 9 abaixo, as amostras de controle agruparam-se muito próximas daquelas alimentadas com PS e farelo, enquanto as dietas de PS solto foram agrupadas juntas.

Figura 8: Medida de diversidade alfa usando o índice de Shannon para larvas de farinha alimentadas com os diferentes tratamentos dietéticos.

Figura 9: Gráfico PCoA bidimensional da diversidade beta com base no índice de dissimilaridade de Bray Curtis entre os gêneros de bactérias entre larvas de farinha alimentadas nos três grupos dietéticos.
Discussão
O poliestireno é um dos tipos de plástico mais comuns usados globalmente, mas sua resiliência à degradação natural resultou em seu acúmulo no meio ambiente7,8,29. Portanto, identificar métodos sustentáveis alternativos para acelerar a biodegradação seria eficaz como estratégia de gerenciamento de resíduos plásticos. Foi comprovado que os insetos têm a capacidade de consumir, degradar e mineralizar plásticos e seu microbioma intestinal foi sugerido para desempenhar um papel importante em seus processos de digestão10,11,30,31. Aqui, apresentamos um estudo de relatório na África sobre a capacidade potencial de uma larva de larva de farinha menor recém-identificada (Coleoptera: Tenebrionidae) de consumir poliestireno e as comunidades microbianas associadas quando alimentadas com dietas PS.
A primeira etapa deste estudo de pesquisa foi fornecer um relatório de identificação usando o perfil mitogenômico da larva da farinha menor coletada no Quênia e apresentar sua relação filogenética. O mitogenoma circular tinha 15.509 pb de comprimento, um pouco mais curto do que os mitogenomas de Alphitobius publicados anteriormente32. Essas diferenças entre os mitogenomas de Alphitobius publicados e a recém-identificada larva de farinha menor sugerem que pode ser uma nova subespécie ou uma nova espécie que requer identificação adicional. A orientação e a ordem dos genes foram idênticas a outros mitogenomas disponíveis publicamente para espécies da família Tenebrionidae e também ao tipo comum sugerido como ancestral dos insetos33,34. O tamanho dos genes codificadores de proteínas era semelhante ao de outros membros da família Tenebrionidae. Todos os tRNAs foram dobrados em uma estrutura típica de folha de trevo, exceto o tRNASor que não possuía um braço de di-hidroudina (DHU) e essa estrutura incompleta também foi detectada em outros grupos de insetos35,36,37.
Nosso estudo revelou que a larva da farinha menor pode ser capaz de consumir poliestireno, como evidenciado pelo consumo de PS e mastigação do isopor. As taxas de consumo de PS aumentaram ao longo do período de 30 dias, mais ainda quando uma co-dieta de farelo foi adicionada. Esses resultados se comparam aos apresentados anteriormente em outras larvas de farinha comuns (T. molitor e T. obscurus), pois a adição de uma co-dieta rica em nutrientes, como o farelo, aumenta o consumo de PS38,39. As taxas de sobrevivência em todos os três grupos de dieta em nosso estudo variaram entre 80 e 100% durante todo o período de alimentação. Um estudo feito por13 capacidade dos super vermes de degradar o PS mostrou uma taxa de sobrevivência de 95% quando alimentados com dietas PS enquanto12 compararam as taxas de sobrevivência de larvas de farinha amarelas e dietas PS alimentadas com traças de cera maiores e descobriram que as taxas de sobrevivência são inferiores a 75%. Essa variabilidade nas taxas de sobrevivência em estudos anteriores e até mesmo em nosso estudo pode ser devido a fatores ambientais, condições de criação e outros. A partir de nosso estudo, as taxas de sobrevivência em larvas alimentadas com linguado PS diminuíram ao longo do tempo, sugerindo que o PS sozinho é uma dieta pobre e uma co-alimentação com farelo é mais viável. As diversidades microbianas em larvas alimentadas com PS e farelo co-alimentadas foram maiores em comparação com aquelas alimentadas apenas com PS. Isso também demonstra que a diversidade microbiana intestinal é muito influenciada pela dieta.
A análise da abundância microbiana relativa revelou que o intestino da larva da farinha menor era dominado pelos filos Proteobacteria e Firmicutes. Outros filos, incluindo Cyanobacteria, Actinobacteria e Bacteroidetes, também estavam presentes, mas em porções menores. Esta observação se alinha com estudos anteriores sobre larvas de farinha comuns, sendo Firmicutes e Proteobacteria os filos dominantes40,41,42, embora em nosso estudo as Proteobactérias tenham sido mais dominantes do que as Firmicutes. As famílias Enterobacteriaceae, Streptococcaceae, Xanthomonadaces e Enterococcaceae foram predominantes em todos os grupos de dieta e essas famílias também foram observadas em outros estudos anteriores envolvendo a degradação do PS por larvas de farinha9,39. Em nosso estudo atual, observamos que a ingestão de PS aumentou a abundância da família Enterobacteriaceae, tornando esse grupo de bactérias os candidatos mais prováveis a estarem envolvidos na quebra inicial do PS no intestino da larva da farinha. A família Enterobacteriaceae é conhecida por suas diversas capacidades metabólicas, incluindo a degradação de vários compostos orgânicos complexos43. Bactérias desta família, incluindo os gêneros Citrobacter, Kosakonia, Enterobacter, Lactococcus e Kluyvera, foram relatadas anteriormente como associadas à degradação do PS e são conhecidas por abrigar enzimas capazes de quebrar polímeros sintéticos38,44,45. O aumento na família Enterobacteriaceae observado em nosso estudo pode ser atribuído à presença de potenciais micróbios degradadores de PS, como Kluyvera, Klebsiella e Citrobacter, indicando uma resposta adaptativa ao poliestireno auxiliando sua quebra e possivelmente degradação adicional.
Uma análise relativa adicional em nível de gênero em nosso estudo indicou que anaeróbios facultativos, γ-proteobactérias, da família Enterobacteriaceae, incluindo os gêneros Kluyvera, Klebsiella e Citrobacter, foram os táxons de bactérias mais abundantes nos grupos de dieta PS. Esses táxons são algumas das bactérias comuns associadas ao intestino em larvas de farinha e insetos em geral. Kluyvera, que era o gênero predominante nas larvas alimentadas exclusivamente com dietas PS, foi identificado como um micróbio potencialmente degradador de PS e polipropileno, embora não tenha sido comumente relatado como uma bactéria degradadora de plástico20. O gênero Lactococcus da família Streptococcaceae também foi observado em alta abundância nas dietas PS em comparação com as dietas controle que continham apenas farelo de trigo. Esta bactéria gram-positiva é conhecida principalmente por seu uso como probiótico e, embora seu papel na degradação do plástico não tenha sido extensivamente estudado, alguns estudos relataram a presença de Lactococcus como uma bactéria degradadora de PS no intestino de Tenebrio molitor46,47. O controle (farelo de trigo), por outro lado, foi dominado pelo gênero Stenotrophomonas da família Xanthomonadaceae, e isso não foi relatado em estudos anteriores, especialmente aqueles que utilizaram o farelo como fonte nutricional. Este gênero é uma bactéria gram-negativa incomum que foi denotada como um patógeno potencial ou comensal de certas espécies, incluindo insetos48. No entanto, um estudo feito por49 que isolaram S. maltophilia de detritos plásticos encontrados no solo sugeriram que esse gênero também poderia ter o potencial de degradar o polietileno.
Numerosos estudos de pesquisa até agora identificaram micróbios degradadores de PS do intestino de insetos. Yang et al.19 foi um dos primeiros estudos a identificar a bactéria degradadora de PS Exiguobacterium sp. YT2 do intestino de T. molitor. Um estudo feito por38 identificou espécies de bactérias intestinais associadas à degradação de PS-, incluindo Citrobacter, Klebsiella e Serratia. Wang et al.50 também investigou micróbios intestinais do intestino de T. castaneum alimentado com dietas PS e foi capaz de identificar Acinetobacter sp. estar altamente associado à degradação de PS. Além disso44, isolado de PE degradando Enterobacter absuriae YT1 das entranhas da traça da farinha indiana e51 PS isolado degradando Pseudomonas sp. do intestino das larvas de Zophobas atratus. O contraste nas comunidades microbianas identificadas nos diferentes estudos destaca os efeitos das composições das dietas e das co-dietas utilizadas principalmente sobre os insetos. Também sugere que os insetos podem conter enzimas digestivas e biorreagentes para a degradação do plástico em seus intestinos e que a degradação do plástico por insetos é uma reação sinérgica ou simbiótica entre os micróbios intestinais e o hospedeiro. Portanto, a pesquisa sobre o microbioma intestinal de insetos que degradam o plástico é uma abordagem importante para entender os insights da biodegradação do plástico por insetos. O papel dos micróbios intestinais na degradação do PS e na biorremediação plástica ainda não é totalmente compreendido e isso deve ser investigado por meio de isolamento e testes enzimáticos para determinar as vias microbianas envolvidas na despolimerização dos plásticos.
Conclusão
Nosso estudo foi capaz de identificar comunidades microbianas do intestino da recém-identificada larva da farinha menor quando alimentada com dietas de poliestireno e a análise completa do mitogenoma forneceu informações valiosas sobre a composição genética, lançando luz sobre suas relações filogenéticas. A presença de micróbios intestinais como Kluyvera, Klebsiella e Lactococcus, possivelmente associados à degradação do PS, apóia relatos feitos por estudos anteriores sobre a capacidade das larvas de farinha de degradar o poliestireno. Nosso estudo gerou novas questões, como quais genes são expressos durante a degradação do PS e quais são as vias completas usadas pelos micróbios. Quais membros da comunidade intestinal estão envolvidos em todo o processo de degradação? À medida que tais questões surgem, o emprego da transcriptômica para quantificar os níveis e funções de expressão gênica contribuirá para resolvê-las. Uma sequência abrangente do mitogenoma servirá como um recurso crucial para estudos futuros que ajudarão na identificação de genes específicos e vias metabólicas envolvidas na degradação do plástico. Além disso, a mitogenômica comparativa pode ajudar na compreensão das adaptações evolutivas das larvas de farinha aos polímeros sintéticos, fornecendo uma perspectiva mais ampla sobre o significado ecológico e funcional da degradação do plástico.
Embora as diversas capacidades das larvas de farinha menores ofereçam possibilidades promissoras no desenvolvimento de estratégias sustentáveis de gerenciamento de resíduos, desafios como otimização, ampliação para aplicação prática e garantia da segurança dos produtos finais precisam ser abordados. Além disso, a eficácia da larva da farinha menor na degradação de diferentes tipos de plásticos e os impactos potenciais em sua saúde e comportamento requerem uma investigação mais aprofundada, bem como sua capacidade de converter resíduos em biomassa rica em proteínas de insetos de alto valor para ração animal. A pesquisa contínua e os esforços colaborativos entre cientistas, formuladores de políticas e indústrias serão fundamentais para realizar todo o potencial das larvas de farinha e outros organismos semelhantes. Esses esforços combinados são a chave para lidar com os resíduos plásticos, ao mesmo tempo em que fornecem proteína de insetos de alto valor para promover uma estrutura de economia circular.
Disponibilidade de dados
Os dados de sequência que apóiam os achados deste estudo foram depositados no NCBI com o número do BioProject PRJNA1102172 e podem ser acessados usando este link.
Fonte: Springer Nature | Autor correspondente: Fathiya M. Khamis.
Autores da publicação: Evalyne W. Ndotono, Chrysantus M. Tanga, Segenet Kelemu & Fathiya M. Khamis
Tradução: Google Tradutor
Publicação original:
Perfil mitogenômico e análise microbiana intestinal da recém-identificada larva da farinha menor consumidora de poliestireno no Quênia
Referências
Perspectivas Globais de Plásticos. Perspectivas Globais de Plásticos (OCDE, 2022). https://doi.org/10.1787/de747aef-en.
Balabantaray, S. R., Singh, P. K., Pandey, A. K., Chaturvedi, B. K. & Sharma, A. K. Previsão da produção global de plástico e emissão de microplásticos usando modelo cinza discreto otimizado avançado. Environ. Sci. Pollut. Res. 30(59), 123039–123054 (2023).
Amankwa, M. O., Tetteh, E. K., Mohale, G. T., Dagba, G. & Opoku, P. A produção de produtos valiosos e combustível a partir de resíduos plásticos na África. Discov. Sustentar. https://doi.org/10.1007/s43621-021-00040-z (2021).
África, S. Identificando lacunas e oportunidades de políticas de poluição plástica na África. www.wwf.org.za/report/plastic_pollution_policy_in_africa (2022).
Associação de Fabricantes de Plásticos. Plásticos – os fatos. Plásticos Europa 38 (2018).
Europa, P. Plastics Europe Associação de Fabricantes de Plásticos Plásticos – os fatos 2021 uma análise dos dados europeus de produção, demanda e resíduos de plásticos. Plásticos – os fatos 2021, 34 (2021).
Ho, B. T., Roberts, T. K. & Lucas, S. Uma visão geral sobre a biodegradação do poliestireno e do poliestireno modificado: a abordagem microbiana. Crit. Rev. Biotechnol. 38, 308–320 (2018).
Maharana, T., Negi, Y. S. & Mohanty, B. Artigo de revisão: Reciclagem de poliestireno. Polym. Plast. Eng. Técnico 46, 729–736 (2007).
Peña-Pascagaza, P. M., López-Ramírez, N. A. & Ballen-Segura, M. A. Tenebrio molitor e seu crescimento de bactérias intestinais na presença de poliestireno (PS) como a única fonte de carbono. Univ. Sci. 25, 37–53 (2020).
Mallar, C. N. R. Uma revisão sobre o papel dos insetos e sua microbiota intestinal na biodegradação do plástico. 79 (2020).
Bilal, H., Raza, H., Bibi, H. & Bibi, T. Biodegradação plástica através de insetos e seus micróbios simbiontes: uma revisão. J. Bioresour. Gestão. 8, 95–103 (2021).
Jiang, S., Su, T., Zhao, J. & Wang, Z. Biodegradação de poliestireno por larvas de Tenebrio molitor, Galleria mellonella e Zophobas atratus e comparação de seus efeitos de degradação. Polímeros 13, 3539 (2021).
Sun, J., Prabhu, A., Aroney, STN & Rinke, C. Insights sobre a biodegradação do plástico: composição da comunidade e capacidades funcionais do microbioma do superverme (Zophobas morio) em testes de alimentação de isopor. Microb. Genom. 8, 1–19 (2022).
Sammarco, B. C., Hinkle, N. C. & Crossley, M. S. Biologia e manejo da larva da farinha Alphitobius diaperinus (Coleoptera: Tenebrionidae) em galpões de frangos de corte. J. Integr. Peste. Gestão. https://doi.org/10.1093/jipm/pmad003 (2023).
Cucini, C. et al. Diversidade bacteriana e fúngica no intestino de Alphitobius diaperinus (Insecta: Coleoptera) alimentado com poliestireno. Gene 17–18, 200109 (2020).
Chalup, A. et al. Primeiro relato da mariposa-das-ceras Achroia grisella F. (Lepidoptera: Pyralidae) consumindo polietileno (silo-bag) no noroeste da Argentina. J. Apic Res. 57, 569–571 (2018).
Suresh Kesti, S. & Chandrabanda Thimmappa, S. Primeiro relato de biodegradação de polietileno de baixa densidade por larvas de traça do arroz, Corcyra cephalonica (stainton). Holista. Aproxime-se do ambiente. 9, 79–83 (2019).
Mahmoud, E. A., Al-Hagar, O. E. A. & El-Aziz, M. F. A. Efeito da radiação gama nas bactérias do intestino médio de Plodia interpunctella e seu papel na biodegradação de resíduos orgânicos. Int. J. Trop. Inseto Sci. 41, 261–272. https://doi.org/10.1007/s42690-020-00203-x (2021).
Yang, Y. et al. Biodegradação e mineralização de poliestireno por larvas de farinha comedoras de plástico: Parte 1. Caracterização química e física e ensaios isotópicos. Environ. Sci. Technol. 49, 12080–12086 (2015).
Yang, S. S. et al. Biodegradação de resíduos de poliestireno em larvas de farinha amarelas (larvas de Tenebrio molitor Linnaeus): Fatores que afetam as taxas de biodegradação e a capacidade das larvas alimentadas com poliestireno de completar seu ciclo de vida. Quimiosfera 191, 979–989 (2018).
Meng, G., Li, Y., Yang, C. & Liu, S. MitoZ: Um kit de ferramentas para montagem, anotação e visualização do genoma mitocondrial animal. Ácidos nucléicos Res. 47, e63 (2019).
Kumar, S., Stecher, G., Li, M., Knyaz, C. & Tamura, K. MEGA X: Análise genética evolutiva molecular em plataformas de computação. Mol. Biol. Evol. 35, 1547–1549 (2018).
Wang, Y., Zhao, Y., Bollas, A., Wang, Y. & Au, K. F. Tecnologia de sequenciamento de nanoporos, bioinformática e aplicações. Nat. Biotechnol. 39, 1348–1365. https://doi.org/10.1038/s41587-021-01108-x (2021).
Bolyen, E. et al. Ciência de dados de microbioma reprodutível, interativa, escalável e extensível usando QIIME 2. Nat. Biotechnol. 37, 852–857 (2019).
Bolger, A. M., Lohse, M. & Usadel, B. Trimmomatic: Um aparador flexível para dados de sequência Illumina. Bioinformática 30, 2114–2120 (2014).
McMurdie, P. J. & Holmes, S. Phyloseq: Um pacote R para análise interativa reproduzível e gráficos de dados do censo do microbioma. PLoS UM 8, e61217 (2013).
Equipe principal R. R: Uma linguagem e ambiente para computação estatística (R Foundation for Statistical Computing, 2023).
Kaufmann, J. & Schering, A. G. Análise de Variância ANOVA.
Lear, G. et al. Plásticos e o microbioma: impactos e soluções. Environ. Microbiomas 16, 1–19 (2021).
Kesti, S. S. & Shivasharana, C. T. O papel dos insetos e microrganismos na biodegradação do plástico. Uma revisão abrangente. Int. J. Sci. Res. Biol. Sci. 5, 75–79 (2019).
Liaqat, S. Ecologia microbiana: uma nova perspectiva da degradação do plástico. Puro Appl. Biol. 9, 2138–2150 (2020).
Hong, K. J., Ki, W., Lee, H., Park, J. & Lee, W. O segundo genoma mitocondrial completo de Alphitobius diaperinus Panzer, 1797 (Coleoptera: Tenebrionidae): Investigação de variações intraespecíficas no genoma mitocondrial. Recursos de DNA mitocondrial B. 5, 2997–2999 (2020).
Taanman, J. W. O genoma mitocondrial: estrutura, transcrição, tradução e replicação. Biochim. Biofísica. Acta Bioenerg. 1410, 103–123 (1999).
Tomberlin, J. K. et al. Um levantamento da diversidade bacteriana de estágios sucessivos da mosca soldado negra (Diptera: Stratiomyidae) usando pirosequenciamento de rDNA 16S. J. Med. Entomol. 50, 647–658 (2013).
Liao, F. et al. O genoma mitocondrial completo da minhoca de outono, Hyphantria cunea (Lepidoptera: Arctiidae). Int. J. Biol. Sci. 6, 172–186 (2010).
Li, H. et al. A arquitetura e a sequência completa do genoma mitocondrial de um inseto assassino Agriosphodrus dohrni (Hemiptera: Reduviidae). Int. J. Biol. Sci. 7, 792–804 (2011).
Liu, L. N. & Wang, C. Y. Genoma mitocondrial completo do verme da farinha amarela (Tenebrio molitor). Dong wu xue yan jiu = Pesquisa zoológica / ‘Dong wu xue yan jiu’ bian ji wei yuan hui bian ji 35, 537–545 (2014).
Brandon, AM et al. Biodegradação de misturas de polietileno e plástico em larvas de farinha (larvas de Tenebrio molitor) e efeitos no microbioma intestinal. Environ. Sci. Technol. 52, 6526–6533 (2018).
Peng, B. Y. et al. Biodegradação do poliestireno por larvas de farinha escuras (Tenebrio obscurus) e amarelas (Tenebrio molitor) (Coleoptera: Tenebrionidae). Environ. Sci. Technol. 53, 5256–5265 (2019).
Wang, Y. & Zhang, Y. Investigação de bactérias associadas ao intestino em larvas de tenebrio molitor (Coleoptera: Tenebrionidae) usando métodos dependentes de cultura e DGGE. Ann. Entomol. Soc. Am. 108, 941–949 (2015).
Cassone, B. J., Grove, H. C., Elebute, O., Villanueva, S. M. P. & LeMoine, C. M. R. Papel do microbioma intestinal na degradação do polietileno de baixa densidade por larvas de lagarta da mariposa da cera maior, Galleria mellonella. Proc. R. Soc. B Biol. Sci. 287, 9–11 (2020).
Jiang, S., Su, T., Zhao, J. & Wang, Z. Isolamento, identificação e caracterização de bactérias degradadoras de poliestireno do intestino de larvas de Galleria mellonella (Lepidoptera: Pyralidae). Frente. Bioeng. Biotecnologia. 9, 1–9 (2021).
Octavia, S. & Lan, R. A família Enterobacteriaceae. Em Os procariontes (eds Rosenberg, E. et al.) 225–286 (Springer, 2014).
Yang, J., Yang, Y., Wu, W. M., Zhao, J. & Jiang, L. Evidência de biodegradação do polietileno por cepas bacterianas das entranhas de vermes comedores de plástico. Environ. Sci. Technol. 48, 13776–13784 (2014).
Yang, Y. et al. Biodegradação e mineralização do poliestireno por larvas de farinha comedoras de plástico: Parte 2. Papel dos microrganismos intestinais. Environ. Sci. Technol. 49, 12087–12093 (2015).
Urbanek, A. K., Rybak, J., Wróbel, M., Leluk, K. & Mirończuk, A. M. Uma avaliação abrangente da diversidade do microbioma em Tenebrio molitor alimentado com resíduos de poliestireno. Environ. Poluente. 262, 114281 (2020).
Przemieniecki, S. W., Kosewska, A., Ciesielski, S. & Kosewska, O. Alterações no microbioma intestinal e perfil enzimático de larvas de Tenebrio molitor biodegradando resíduos de celulose, polietileno e poliestireno. Environ. Poluente. 256, 113265 (2020).
Brooke, J. S. Avanços na microbiologia de Stenotrophomonas maltophilia. Clin. Microbiol. Rev. https://doi.org/10.1128/CMR.00030-19 (2021).
Peixoto, J., Silva, L. P. & Krüger, R. H. O solo do Cerrado brasileiro revela um potencial microbiano inexplorado para a biodegradação de polietileno não pré-tratado. J. Perigo Mater. 324, 634–644 (2017).
Wang, Z., Xin, X., Shi, X. & Zhang, Y. Bactéria Acinetobacter degradadora de poliestireno isolada das larvas de Tribolium castaneum. Sci. Ambiente Total. 726, 138564 (2020).
Kim, H. R. et al. Biodegradação do poliestireno por Pseudomonas sp. Isolado do intestino de supervermes (larvas de Zophobas atratus). Environ. Sci. Technol. 54, 6987–6996 (2020).












































